新型コロナウイルスゲノム解析
新型コロナウイルス感染症は、その感染力と死亡率の高さから公衆衛生上極めて重要な疾病と認識されています。医療崩壊の危機が迫る中で、新型コロナウイルスゲノム配列情報を利用した新しい診断法と感染予防体制の構築が喫緊事です。慶應義塾大学医学部臨床遺伝学センターは、国立研究開発法人日本医療研究開発機構(AMED)の支援により、「COVID-19ウィルスゲノムシーケンシングによるワクチン・薬剤耐性関連変異株・海外変異株の予防的国内監視システムの構築」を推進しています。
協力医療機関より患者様の検査残余検体及び臨床情報を収集するウイルスゲノムネットワークを構築、さらにウイルス全ゲノム解析プロトコルの最適化を図り、種々の検査残余検体から抽出したウイルスRNAの全ゲノム配列を高精度で解析することに成功しました。解析により得られた新規ゲノム配列情報は、積極的に国際データベースに登録・公開し、全世界の公衆衛生研究発展に寄与します。
解析した新型コロナイルスのゲノム配列情報は感染ルートを正確に把握するための判断の一助となります。感染の伝搬の有無や経路を科学的に決定するシステムを構築・活用することで、本感染症による院内感染の拡大回避・医療体制の早期回復、及び医療従事者の負担の軽減が期待されます。また、ゲノム配列情報から得られたウイルス変異に臨床情報を突合することで、新型コロナウイルスの病原性や感染性などのメカニズムを解明し、本感染症の診断、治療、感染対策への貢献を目指します。
最新情報
 新型コロナウィルスSARS-Cov-2ゲノム配列の一般公開を開始
新型コロナウィルスSARS-Cov-2ゲノム配列の一般公開を開始新型コロナウィルスSARS-Cov-2ゲノム配列の一般公開を開始いたしました。 これまでG...
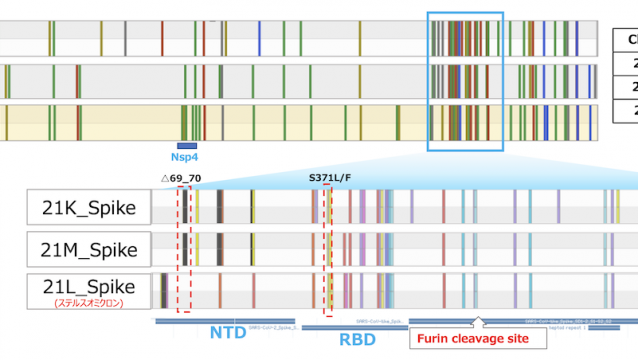
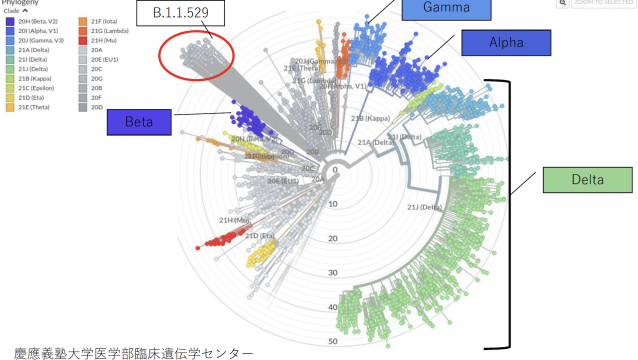
 日本国内新型コロナウィルス変異分子系統樹情報(2021年5月作成)
日本国内新型コロナウィルス変異分子系統樹情報(2021年5月作成)Nextstrainは、病原体ゲノムデータの科学的・公衆衛生的可能性を引き出すためのオープ...
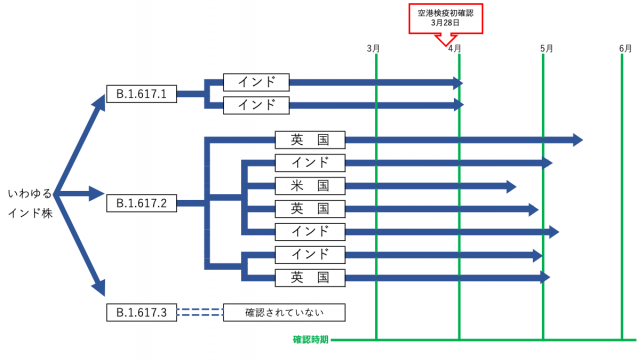
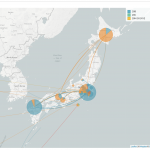
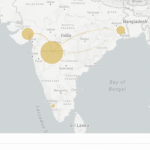 インド国内における、B.1.617(いわゆる3重変異株)の拡散状況の可視化
インド国内における、B.1.617(いわゆる3重変異株)の拡散状況の可視化国際SARS-CoV-2のゲノム配列データベースGISAIDのインドのデータをインドCSI...
 再度、日本特有株B.1.1.214の中でS:E484Kを有する株が発生
再度、日本特有株B.1.1.214の中でS:E484Kを有する株が発生われわれは2021年3月4日付で、日本に特有の株であるB.1.1.214に属する株の中でE...
 米国西海岸由来株が3か月間で最初の株から150株に増加
米国西海岸由来株が3か月間で最初の株から150株に増加われわれは、2021年1月12日に、米国西海岸から日本に20Cに分類されるB.1.346株...
 免疫逃避変異E484Kを含むR1株の国内分布
免疫逃避変異E484Kを含むR1株の国内分布わが国においてスパイクタンパクに免疫逃避変異とされるE484Kを有するR.1株が初めて検出...
関連報道記事・番組
研究成果
- Identification of SARS-CoV-2 Clade 20C in Japan (November, 2020) (2021/01/12)
The whole genome sequence of the starin has been deposited to the GISAID database (hCoV-19/Japan/Donner96/2020) on January 12, 2021.
Identification of B.1.346 lineage of SARS-CoV-2 in Japan: Genomic evidence of re-entry of Clade 20C (2021/02/01 medRxiv) - Severity of COVID-19 is inversely correlated with increased number counts of non-synonymous mutations in Tokyo (2020/11/24 medRxiv)
- Clinical utility of SARS-CoV-2 whole genome sequencing indeciphering source of infection (2020/10/04 The Journal of Hospital Infection )
研究事業: 国立研究開発法人日本医療研究開発機構(AMED)
- 「COVID-19ウィルスゲノムシーケンシングによるワクチン・薬剤耐性関連変異株・海外変異株の予防的国内監視システムの構築」
- 「ウイルス全ゲノムシーケンシングを用いた院内感染拡大防止支援システムの基礎研究」
Variant calling pipeline for amplicon-based sequencing of the SARS-Cov-2 viral genome
Overview of the analytic pipeline “Donner” for variant calling from amplicon-based sequencing of the SARS-Cov-2 viral genome sequences.